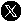News Lexikon
Durchbruch bei Entschlüsselung von Tollwut
- Details
- Hauptkategorie: News Lexikon
- Kategorie: Gesundheit, Medizin & Wellness NEWS
- Erstellt: Donnerstag, 05. September 2019 18:22
- Zuletzt aktualisiert: Donnerstag, 05. September 2019 18:22
- Veröffentlicht: Donnerstag, 05. September 2019 18:22
- Geschrieben von Super User
- Zugriffe: 1008
Durchbruch bei Entschlüsselung von Tollwut
Wissenschaftler der Vetmeduni Vienna gehen Struktur des Ribonukleoproteins auf den GrundKryoelektronentomografie genutzt
Das RABV-Partikel besteht aus einer von Zellen abgeleiteten Membran, in der mehrere Kopien des sogenannten Oberflächenglykoproteins verankert sind, und einem helikalen Ribonukleoprotein (RNP), das an einem Ende eine konische Spitze bildet. Obwohl die einzelnen Bauteile des RNP bereits bekannt waren, konnte die exakte Struktur des intakten RABV-RNP-Komplexes bisher nicht bestimmt werden. Genau das ist nun jedoch einem Team der Vetmeduni Vienna gelungen.
Mittels Kryoelektronentomografie - einem Verfahren zur Darstellung biologischer Strukturen - und einer daran anschließenden computerunterstützten Analyse haben die Forscher den Aufbau des Virus analysiert. Demnach besteht die Virusstruktur aus einer rechtshändigen Helix, die am 3'-Ende des Genoms im RNP-Konus lokalisiert ist, also gleich wie beim verwandten Virus der vesikulären Stomatitis (VSV), einer mild verlaufenden Viruskrankheit, die vor allem bei Huftieren auftritt und beim Menschen zu grippeähnlichen Symptomen führen kann.
"Erstaunliche strukturelle Variabilität"
"Die Verbindung benachbarter Helix-Windungen erfolgt beim Tollwutvirus allerdings über eine Interaktion des M-Proteins mit dem N-Protein, während eine M-M-Interaktion für VSV beschrieben wurde. Daraus resultiert - im Vergleich zu VSV - auch ein größerer Abstand der Helixwindungen zueinander und ein flacherer Winkel der einzelnen RNP-Windungen zur zentralen Virusachse", verdeutlicht Erstautorin Christiane Riedel.
Der Expertin nach demonstriert dies eine "erstaunliche strukturelle Variabilität des RNPs zwischen VSV und RABV", obwohl die schon bestimmten Kristallstrukturen der Einzelbausteine, also der N- und M-Proteine, strukturell stark homolog sind. Oder anders gesagt: "Obwohl die Einzelbausteine des Tollwutvirus und des VSV sehr ähnlich sind, unterscheiden sich die RNPs der beiden Viren deutlich in ihrer Architektur", so Riedel zu den zentralen Studienergebnissen.
Aussender: pressetext.redaktion
Ansprechpartner: Florian Fügemann
E-Mail: Diese E-Mail-Adresse ist vor Spambots geschützt! Zur Anzeige muss JavaScript eingeschaltet sein!
Tel.: +43-1-81140-313
Website: www.pressetext.com
© pressetext.deutschland +++ pressetext.austria +++ pressetext.schweiz +++ termindienst +++ fotodienst +++ newsfox.com und der jeweilige Aussender
http://www.pressetext.com/news/20190729021pte20190729021
Forschung/Technologie, Medizin/Wellness
Medieninhaber und Herausgeber:
pressetext Nachrichtenagentur GmbH, Josefstädter Straße 44, A-1080 Wien
pressetext ist reichweitenstärkster Nachrichtenverbreiter für Entscheider und Journalisten in der DACH-Region. Die inhaltliche Verantwortung für redaktionelle Meldungen (pte) liegt bei pressetext, für Pressemitteilungen (pts) und Kapitalmarktmitteilungen börsennotierter Unternehmen (pta) beim jeweiligen Aussender. Die Nachrichten werden auf den pressetext-Länderplattformen publiziert und je nach Abonnement-Profil und gewählter Zustellart einzeln oder täglich als Newsletter an die Abonnenten verschickt. Weitere Informationen erhalten Sie bei unserem Redaktionsservice unter Tel. +43-1-81140-300.